multivariate_normalを使って6個のデータポイント近隣にデータ達を発生させる。
薄くクラスタを見つけられそうだけれど境界は曖昧で大分被っているという状況。
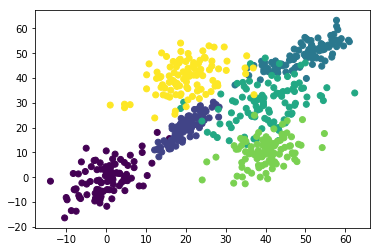
そんなデータ達に6-meansをかけてみたとき、どうクラスタが出来るのかという実験。
被っている部分がどう別のクラスタに入るのか確認するため。
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
mu = [[0,0], [20,20], [50,50], [40,30], [40,10], [20,40]]
sigma = [ [[30,20],[20,50]], [[20,30],[10,20]], [[60,40],[20,20]], [[60,20],[20,60]] ,[[30,10],[10,30]],[[50,20],[20,50]] ]
points = 100
clusteres = []
for index in range(len(mu)):
cluster = np.random.multivariate_normal(mu[index], sigma[index], points)
dig = np.full((points,1),index+1, dtype=int)
cluster = np.hstack((cluster,dig))
clusteres = np.r_[clusteres,cluster] if len(clusteres) > 0 else cluster
plt.scatter(x=clusteres[:,0], y=clusteres[:,1],c=clusteres[:,2])
おもむろにsklearnのKMeansを使ってみる。
n_clustersは6、max_iterを10に設定してみた。
クラスタ中心がわかるように重ねてみた。
from sklearn.cluster import KMeans
kmeans_model = KMeans(n_clusters=6, init='random',max_iter=10).fit(clusteres[:,:2])
labels = kmeans_model.labels_
centers = kmeans_model.cluster_centers_
centers
fig = plt.figure()
ax1 = fig.add_subplot(1,1,1)
ax1.scatter(x=clusteres[:,0], y=clusteres[:,1],c=labels)
ax2 = fig.add_subplot(1,1,1)
ax2.scatter(x=centers[:,0], y=centers[:,1], alpha=0.5,s=600,c="pink",linewidth=2,edgecolors="red")

当然のごとく、微妙に被っていた部分は異なるクラスタに分類された。
今回は当たり前なことを確認して終了。
2次元データじゃつまらない…。