クラスタリングについて凄まじくわかりやすい説明を聞いて感動しました。
k-meansを自力で書くことで理解が深まりそうなので、参考にしながらアプトプットしてみます。
前回のエントリで、多次元正規分布に従う標本を作れるようになったので、
もともと存在する中心から適当に散らばるように作ったデータポイントに対して、
どの程度クラスタ分割できるのか確認する例です。
見てわからないと仕方がないので2次元とします。
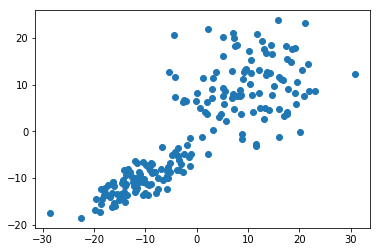
早速、2つの中心から適当に散らばるようなデータポイントを作ってみます。
cluster1 = np.random.multivariate_normal([-10,-10], [[20,10],[30,20]], 100)
cluster2 = np.random.multivariate_normal([10,10], [[50,5],[5,50]], 100)
X = np.r_[cluster1, cluster2]
plt.scatter(X[:,0], X[:,1])
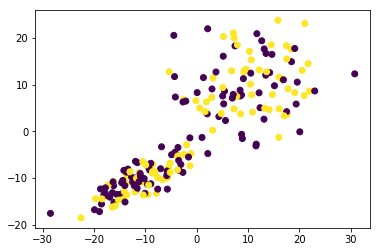
合計200個の点に、確率0.5でラベル1を振って(=確率0.5でラベル0)、
それぞれ色を付けて表示してみます。これを初期クラスタとします。
0クラスタ、1クラスタそれぞれ混じっていて、このままでは適切なクラスタ分割ではありません。
y = np.random.binomial(n = 1, p = 0.5, size = 200)
plt.scatter(X[:,0], X[:,1],c=y)
ラベル0のデータをX1、ラベル1のデータをX2という風に名前を付けて、
pandasデータフレーム化します。
さらに振ったラベル列を付けます。列追加は.assign()で出来るみたい。
import pandas as pd
center = pd.DataFrame(X, columns = ["X1", "X2"])
center.shape # (200, 2)
center = center.assign(y=y)
center.shape # (200, 3)
0クラスタと1クラスタでまとめます。SQLで言うところのGroupBy句。
メソッド名もまんまgroupbyなのでわかりやすい。
GroupByした後、平均を計算する。0クラスタ、1クラスタ毎のX1,X2の平均が出ているっぽい。
center = center.groupby(y).mean()
# X1 X2 y
# 0 -1.701594 -1.186065 0
# 1 1.278607 0.954203 1
いよいよ、各データポイントのクラスタを更新して、適切なクラスタに振っていきます。
pandasデータフレームからNumPyのArrayを取るのは結構面倒くさい。
locにより行を取得できる。その際pandasの列名を指定できる。
SQLでSELECT書いているのに近い。
クラスタ0、クラスタ1それぞれのデータポイントについて、
各クラスタにおけるクラスタ中心と距離を求めます。距離は2乗和。
center_cluster_0 = center.loc[0, ["X1", "X2"]].values
center_cluster_1 = center.loc[1, ["X1", "X2"]].values
center0 = center.loc[0,["X1","X2"]].values
center1 = center.loc[0,["X1","X2"]].values
dist0 = (X[:,0].reshape(200) - center0[0])**2 + (X[:,1].reshape(200) - center0[1])**2
dist1 = (X[:,0].reshape(200) - center1[0])**2 + (X[:,1].reshape(200) - center1[1])**2
クラスタ0のクラスタ中心との距離よりも、クラスタ1のクラスタ中心との距離の方が大きいということは
クラスタ0に属するべきです。論理式の結果がTrueかFalseかのいずれかになることを使って
2つのクラスタに分けます。かなり無理やりな気がします。
真偽の扱いですが、Pythonの言語仕様上、0か0に類する値はFalse、それ以外はTrueです。
new_cluster_id = dist0 < dist1
元Webプログラマが普通にforを使って書いてみると以下の通りとなります。
new_cluster_id2 = []
for index in range(len(dist0)):
new_cluster_id2.append( 0 if (dist0[index] < dist1[index]) else 1)
np.reshape(new_cluster_id2,200)
生成した入力値に新しいクラスタIDを振って色分け表示してみます。
new_cluster_id = dist0 < dist1
plt.scatter(X[:,0], X[:,1], c = new_cluster_id)
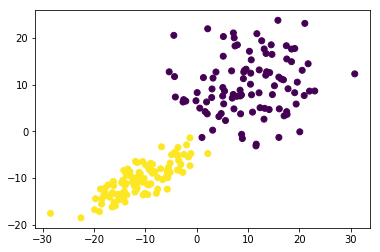
綺麗にわかれた気がしますが、大元のクラスタが交差する部分において、
別のクラスタに分類されたデータがあるようです。