k-means法を実行する際に妥当なkを決めたいという欲求があります。
クラスタ集合の凝集度を定量化することでkと凝集度の関係を得られます。
複数のkについてkと凝集度の関係を取得し、
そこから妥当なkを発見する方法がエルボー法です。
この記事においては以下を書いてみます。
- クラスタ集合の凝集度を定量化する方法
- kと凝集度の関係を表すグラフの書き方
クラスタ集合の凝集度を定量化する方法
これ以上無いくらいシンプルなやり方です。
データポイントとcentroidの距離の2乗和(SSE)を凝集度として使います。
それを全クラスタに関して計算し足し合わせます。
足し合わせた値を歪み(distortion)と言っているところが多いです。
クラスタ数kが増えるにつれてこの値は小さくなるだろうと予測できます。
横軸にk、縦軸にSSEの和をとるグラフを書いてみると、
kの増加に伴ってSSEの和が小さくなること、減少幅は次第に小さくなりそうです。
(すべてのケースでそうなるのかは不明)
減少幅が緩やかになる地点のkが費用対効果が高いkですし、
ほぼ減少が止まる(サチる)地点のkを採用することは、
それ以上kを増加させても意味がないという事実を根拠として使えそうです。
擬似データへの適用
前回から使っている多次元正規分布に従う擬似クラスタデータに対してk-meansを適用します。
k=1から9まで適用し、k-distortionグラフを書いてみます。(だいぶPythonに慣れてきた..。)
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
mu = [[0,0], [20,20], [50,50], [40,30], [40,10], [20,40]]
sigma = [ [[30,20],[20,50]], [[20,30],[10,20]], [[60,40],[20,20]], [[60,20],[20,60]] ,[[30,10],[10,30]],[[50,20],[20,50]] ]
points = 100
clusters = []
for index in range(len(mu)):
cluster = np.random.multivariate_normal(mu[index], sigma[index], points)
dig = np.full((points,1),index+1, dtype=int)
cluster = np.hstack((cluster,dig))
clusters = np.r_[clusters,cluster] if len(clusters) > 0 else cluster
plt.scatter(x=clusters[:,0], y=clusters[:,1],c=clusters[:,2])
from sklearn.cluster import KMeans
kmeans_model = KMeans(n_clusters=6, init='random',max_iter=10).fit(clusters[:,:2])
labels = kmeans_model.labels_
distortions = []
numClusters = 10
for i in range(1,numClusters):
kmeans_model = KMeans(n_clusters=i, init='random',max_iter=10)
kmeans_model.fit(clusters[:,:2])
distortions.append(kmeans_model.inertia_)
plt.plot(range(1,numClusters),distortions,marker='o')
plt.xlabel('Number of clusters')
plt.ylabel('Distortion')
plt.show()
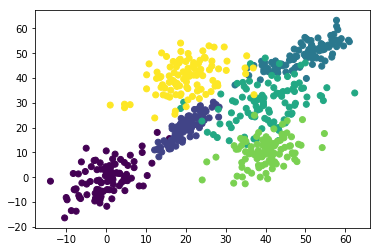
複数の擬似点を中心に2次元正規分布に従う散らばりをもったデータ達です。
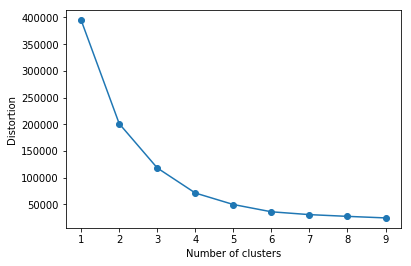
k-distortionグラフです。もの凄い期待通りなグラフができました。
k=5か6あたりでdistortionがサチっています。
6-meansを適用し、centroidを重ねてみました。

散布図を見ての想像になりますが、
確かに4-meansと5-meansでは、5-meansの方が凝集していそうです。
5-meansと6-meansだと、4と5程凝集度合いに変化がなさそうです。